Determinare il sierotipo di Actinobacillus pleuropneumoniae (App) é importante per il suo controllo, poiché diversi sierotipi hanno un diverso potenziale di virulenza (a seconda della zona geografica) e queste informazioni possono essere utilizzate per scegliere il vaccino più appropriato (Gottschalk, 2015). Sulla base della sua capsula, ci sono 18 sierotipi di App noti (Bossé et al., 2018a), essendo 1, 5, 9 e 11 i più virulenti. In base ai requisiti del NAD (nicotinamide adenina dinucleotide), gli isolati del biotipo 1 sono differenziati da quelli del biotipo 2 o NAD-indipendenti.
Classicamente, sono stati utilizzati test sierologici tra cui agglutinazione, coagglutinazione, immunodiffusione, emoagglutinazione indiretta e precipitazione ad anello per determinare il sierotipo. Tuttavia, sono richiesti antisieri specifici con titoli elevati, e le reazioni crociate tra sierotipi (p.e. 3/6/8 e 1/9/11) sono un problema. Abbiamo scoperto che la maggior parte degli isolati del Regno Unito e dell'Irlanda assegnati sierologicamente come sierotipo 3 apparteneva al sierotipo 8 (O'Neill et al., 2010). A causa di questi problemi, abbiamo sviluppato test molecolari utilizzando la reazione a catena della polimerasi (PCR) per amplificare specifiche sequenze di DNA di ciascun sierotipo trovato nei geni di biosintesi della capsula (Bossé et al., 2014). L'uso di queste sequenze specifiche di ciascun sierotipo ha permesso di sviluppare un test specifico per il sierotipo 16 (Bossé et al., 2017) e ha portato alla recente scoperta dei sierotipi 17 e 18 (Bossé et al., 2018a). Con la scoperta dei sierotipi 16-18 abbiamo deciso di produrre un test di PCR che potrebbe assegnare tutti i sierotipi conosciuti di App. Tuttavia, i sierotipi 9 e 11 non possono essere differenziati mediante PCR, poiché i loro loci capsulari sono praticamente identici (Bossé et al., 2018b), né possono essere determinati mediante test sierologici (Gottschalk, 2015). Il test PCR ha dovuto confermare l'isolato come App e determinare il sierotipo specifico. Per confermare l'App, abbiamo usato primer che hanno amplificato una regione di 418 bp del gene apxIV, specifica di App (Schaller et al., 1999). Tuttavia, a causa dell'elevato numero di sierotipi (n = 18), abbiamo dovuto formulare due PCR multiplex (mPCR), ciascuno in grado di rilevare molti sierotipi. L'mPCR1 rileva i sierotipi 1-12 e 15 (Figura 1A) e l'mPCR2 i sierotipi 13-14 e 16-18 (Figura 1B). Gli isolati che amplificano solo una banda apxIV nel mPCR1 sono analizzati dopo con l'mPCR2.

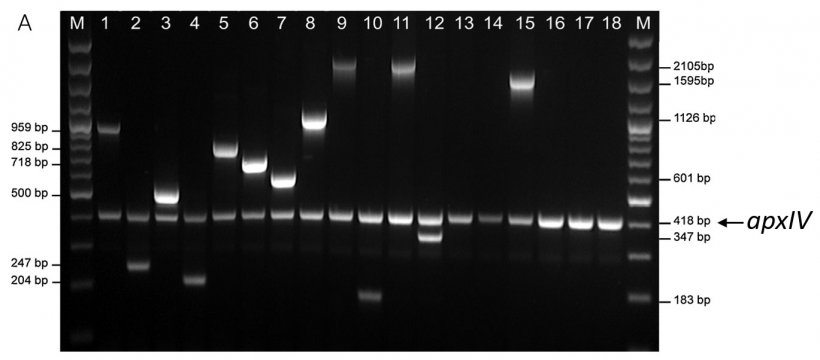
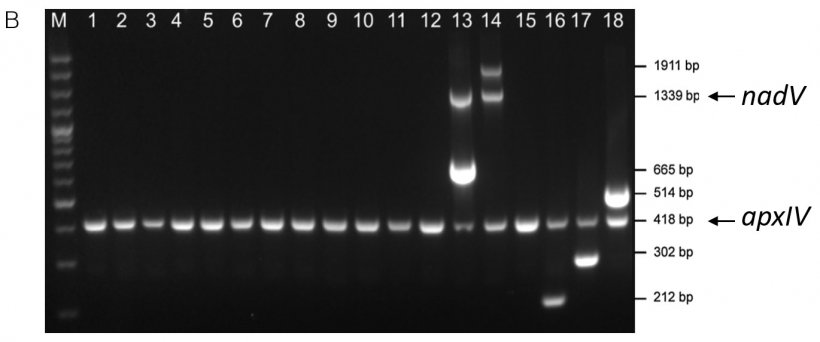
Figura 1. Detección específica de serotipos de amplicones de las cepas de referencia de App por (A) mPCR1 para los serotipos 1-12 y 15 y (B) mPCR2 para los serotipos 13–14 y 16–18. Serotipo de las cepas de referencia 1-18 = líneas 1-18. Ambos mPCR muestran un amplicón apxIV de 418 pb que confirma que las muestras son App (adaptado de Bossé et al., 2018b).
L'mPCR2 conferma anche il biotipo di primer progettati per amplificare un frammento di 1339 bp del gene nadV (che conferisce indipendenza al NAD). L'amplicone nadV di 1339 bp viene rilevato solo nei ceppi di riferimento del biotipo 2 (sierotipi 13-14). Va notato che altri sierotipi (ad esempio 2, 4, 7 e 17) sono stati descritti come appartenenti al biotipo 2 e alcuni sierotipi di isolati nordamericani del sierotipo 13 come appartenenti al biotipo 1 (Gottschalk, 2015).
Il modello di DNA per le PCR può essere il DNA purificato (sia da batteri in coltura o campioni di tessuto) ottenuto utilizzando kit commerciali, lisati di cellule batteriche bollite intere o colonie di una coltura in piastre. Il DNA purificato fornisce risultati migliori, mentre le PCR delle colonie possono dare risultati parziali / falsi negativi se le colonie sono molto appiccicose e difficili da lisare, come dimostrato dai tre isolati clinici del sierotipo 2 che hanno solo amplificato la specifica banda di sierotipi PCR colonia, ma ciò ha amplificato sia questa che la specifica banda di apxIV usando il DNA purificato (Figura 2). In rare occasioni, gli isolati non saranno in grado di amplificare l'apxIV, anche usando il DNA purificato (Bossé et al., 2014). In questo caso, primer apxIV alternativi (oAPXIV-TSP1 / 2) possono essere utilizzati per confermare l'App (Tegetmeyer et al., 2008). mPCR1 e mPCR2 possono essere usati per identificare nuovi sieritipi, come abbiamo fatto con il 17 e 18 (Bossé et al., 2018a). Gli isolati di App che producono una banda apxIV, ma non specifici ampliconi di sierotipo potrebbero essere potenzialmente un nuovo sierotipo, sebbene l'assenza di una specifica banda di sierotipo possa essere dovuta a una mancata corrispondenza del primer in isolati divergenti o a causa della presenza di un elemento di inserimento che altera il locus della capsula. Il sequenziamento del genoma completo dell'isolato confermerà quale di queste opzioni è corretta.
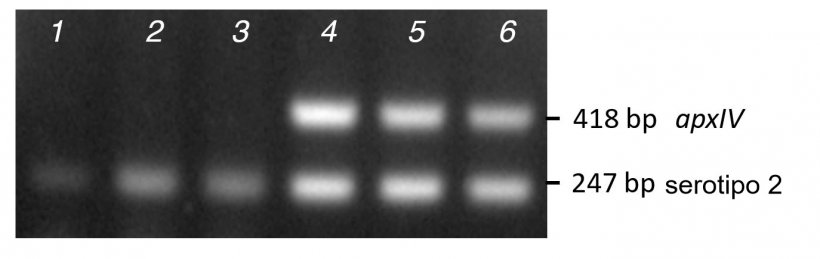
Figura 2. Comparación de la amplificación de bandas a partir de PCR de colonias purificadas (líneas 1-3) versus ADN (líneas 4-6) para tres aislados clínicos de serotipo 2 utilizando el mPCR1.
In sintesi, il controllo dei sierotipi APP in un allevamento o in un paese è importante per controllare la malattia. Le nostre PCR sierotipiche (mPCR1 e mPCR2) sono strumenti utili per identificare i sierotipi virulenti, i vaccini corretti da utilizzare (commerciali o autogeni) e per impedire che suini con ceppi di App potenzialmente virulenti vengano introdotti in allevamenti non esposti. Inoltre, il nostro metodo ha il potenziale per identificare nuovi sierotipi di App e migliorare la diagnostica.
Ringraziamenti
La ricerca sull'App nel laboratorio degli autori è supportata dal Consiglio di ricerca sulle biotecnologie e le scienze biologiche del Regno Unito.




